В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2023-4-43-46
УДК 636.2;4.082
ПОИСК ГЕНЕТИЧЕСКИХ ФАКТОРОВ, СВЯЗАННЫХ С ДЕФЕКТАМИ КОНЕЧНОСТЕЙ У СВИНЕЙ
Е.А. РОМАНЕЦ1, аспирант, С.Ю. БАКОЕВ2, кандидат биолог. наук, вед. аналитик, Л.В. ГЕТМАНЦЕВА2, доктор биолог. наук, вед. аналитик, М.А. КОЛОСОВА1, кандидат с.-х. наук, доцент, e-mail: m.leonovaa@mail.ru, Т.С. РОМАНЕЦ1, кандидат с.-х. наук, ст. преподаватель, Н.А. МАКСИМОВ1, студент факультета ветеринарной медицины, 1ФГБОУ ВО ДГАУ, 2ФГБУ «ЦСП» ФМБА России
Интенсивное производство продукции свиноводства сопряжено с негативным технологическим давлением на организм свиней. Наросты и шишки на задних конечностях (ЗНШ) влияют на внешний вид племенных свиней, и они уже не пригодны к продаже, что крайне негативно сказывается на эффективности работы племенных предприятий.
В статье представлены пилотные исследования, направленные на поиск геномных областей и генов, относящихся к ЗНШ у свиней. Результаты исследований показали 26 SNPs, связанных с ЗНШ у свиней породы ландрас, 15 из них локализованы в генах, задействованных в различных физиологических процессах в организме, в том числе обусловленных воспалениями, различного рода новообразованиями и опухолями.
Ключевые слова: свиньи, наросты, шишки на задних конечностях (ЗНШ), ландрас, SNP, FST, геномные области, ген.
Search for genetic factors associated with limb defects in pigs
E.A. ROMANETS1, postgraduate student, S.Yu. BAKOEV2, candidate of biological sciences, leading analyst, L.V. GETMANTSEVA2, doctor of biological sciences, leading analyst, M.A.KOLOSOVA1, candidate of agricultural sciences, associate professor, e-mail: m.leonovaa@mail.ru, T.S. ROMANETS1, candidate of agricultural sciences, senior lecturer, N.A. MAKSIMOV1, student of the faculty of veterinary medicine, 1Don State Agrarian University, 2Centre for Strategic Planning and Management of Biomedical Health Risks of the Federal Medical Biological Agency)
Intensive production of pig products is associated with negative pressure on the pigs' bodies. Limb outgrowths and bumps affect the appearance of breeding pigs, and they are no longer fit for sale, which has an extremely negative impact on the efficiency of pig farms.
This paper presents pilot studies aimed at finding genomic regions and genes associated with hind limb outgrowths and bumps in pigs. The results showed 26 SNPs associated with hindlimb outgrowths and bumps in Landrace pigs, of which 15 are localized in genes involved in various physiological processes in the body, including those associated with inflammation, various kinds of neoplasms and tumors.
Key words: pigs, hind limb outgrowths and bumps, Landrace, SNP, FST, genomic regions, gen.
Введение
Селекция на скороспелость и мясность приводит к изменению обмена веществ в организме животного, следствием чего являются морфологические и функциональные перестройки внутренних органов, мышечной, жировой и костной ткани. В свиноводстве болезни конечностей влекут за собой большие экономические потери из-за снижения продуктивности животных [1, 2]. Так, например, слабость конечностей (остеохондроз) является источником значительных убытков. Одной из серьезных проблем, с которой столкнулись производители свинины, является образование шишек и наростов на конечностях свиней, которое, как правило, не приводит к хромоте, но влияет на внешний вид племенных свиней, и они уже не пригодны к продаже, что крайне негативно сказывается на рентабельности свиноводческих селекционных центров [3].
В качестве предрасполагающих факторов этиологии ЗНШ у свиней можно рассматривать технологию производства и генетику. Выявление участков генома, прямо или косвенно связанных с ЗНШ, может помочь идентифицировать генетические варианты и использовать их в качестве генетических маркеров при отборе племенного поголовья свиней.
Одним из подходов к поиску генов-кандидатов, ценных с точки зрения селекции признаков сельскохозяйственных животных, является идентификация подписей селекции [10]. Обнаружение подписей селекции и определение генов-кандидатов позволяет установить основные гены, ответственные за выбранные признаки. Преимущество такого подхода заключается в том, что он не зависит от информации о фенотипе отдельных животных и применим к относительно небольшим исследуемым популяциям[6].
Цель исследования ‒ поиск локусов, связанных с ЗНШ у свиней, с помощью метода FST. Этот метод используется для идентификации областей генома при отборе и полезен для анализа популяций, поскольку выявляет тонкие различия между ними.
Материалы и методы исследования
Эксперименты производили на свиньях породы ландрас (n=47) в соответствии с «Правилами проведения работ с использованием экспериментальных животных» (приложение к приказу Министерства здравоохранения СССР от 12.08.1977 года №755).
Свиньи породы ландрас были разделены на две группы в зависимости от состояния конечностей: в первую группу (n=22) вошли животные с наличием, а во вторую (n=25) ‒ с отсутствием шишек в области скакательного сустава задних конечностей.
Генотипирование проводили с помощью GeneSeek® GGP Porcine HD Genomic Profiler v1 (Illumina Inc., США). Для идентификации геномных областей, связанных с ЗНШ свиней, использовали статистику FST путем сравнения генетических вариантов у свиней первой и второй группы. Значимыми вариантами считали те, у которых значения FST превышали уровень квантиля 0,999.
Результаты исследований и их обсуждение
Результаты исследований показали 26 SNPs, связанных с ЗНШ у свиней породы ландрас. Варианты локализованы в 1-й, 4-й, 5-й, 8-й, 10‒14-й, 17-й и 18-й хромосомах (рис.). В 14-й и 10-й хромосоме обнаружено по 4 SNP, в 8-й ‒ 13 и в 17-й ‒ по 3 SNP-варианта (рис.).
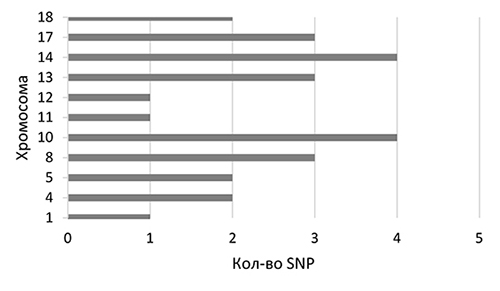
Рис. Распределение обнаруженных SNP по хромосомам
Установленные SNPs были представлены различными вариантами нуклеотидных замен: 1 SNP ‒ синонимичная замена (synonymoys variant), 1 SNP ‒ вариант последовательности, расположенный на 3′-конце гена (downstream gene variant), 11 SNP ‒ межгенные варианты (intergenic variant) и 13 SNP – нуклеотидные замены в интронах (intron variant). Всего в области генома с сильными выбросами определены 15 генов ‒ VAV2, PTPN22, ADORA3, NCF4, LRRIQ1, MARCHF1, ENSSSCG00000060728, SLC43A2, ROBO1, KIF13B, GALNT7, LONRF1, ENSSSCG00000028004, PRKAG2, CREB3L2, задействованных в различных физиологических процессах в организме, в том числе связанные с воспалением, разного рода новообразованиями и опухолями (табл.). Так, VAV2 (vav guanine nucleotide exchange factor 2) участвует в онкогенезе кожи и регулирует количество, функциональный статус и реактивность стволовых клеток выпуклости волосяного фолликула [8]. Ген GALNT7 (polypeptide N-acetylgalactosaminyltransferase7), являясь членом семейства ацетилгалактозаминилтрансфераз, реализует определенный биологический эффект, регулируя взаимодействие между опухолевыми клетками и внеклеточной средой [7]. Ген PRKAG2 (protein kinase AMP-activated non-catalytic subunit gamma 2) предоставляет инструкции для создания одной части (субъединицы гамма-2) более крупного фермента, называемого AMP-активируемой протеинкиназой (AMPK).
Этот фермент помогает ощущать потребность в энергии внутри клеток и реагировать на нее. Он активен во многих различных тканях, включая сердечную мышцу и мышцы, которые используются при движении (скелетные мышцы). Ген ROBO1 (roundabout guidance receptor 1) относится к суперсемейству иммуноглобулинов, представляет собой кольцевой рецептор наведения аксона и имеет широкий спектр, предполагающий разнообразные функции в развитии нервной системы [1, 4].
Таблица 1. Идентифицированные SNP у свиней породы ландрас
|
Хромосома |
Позиция |
Значение FST |
SNP |
Вариант |
Последовательность |
Ген |
|
1 |
273446574 |
0,125 |
G/A |
rs80879386 |
intron variant |
VAV2 |
|
4 |
106740647 |
0,107 |
T/G |
rs3472172726 |
intron variant |
PTPN22 |
|
4 |
108857200 |
0,115 |
T/C |
rs80922198 |
synonymoys variant |
ADORA3 |
|
5 |
10982746 |
0,136 |
T/C |
rs319367356 |
intron variant |
NCF4 |
|
5 |
96886168 |
0,123 |
C/T |
rs329411432 |
intron variant |
LRRIQ1 |
|
8 |
52458798 |
0,108 |
C/T |
rs81400568 |
intron variant |
MARCHF1 |
|
8 |
59670278 |
0,108 |
T/C |
rs3472589911 |
intergenic variant |
|
|
8 |
88163329 |
0,106 |
T/C |
rs335963977 |
downstream gene variant |
ENSSSCG00000060728 |
|
10 |
2653785 |
0,116 |
C/T |
rs328662038 |
intergenic variant |
|
|
10 |
5106897 |
0,135 |
Т/С |
rs333983438 |
intergenic variant |
|
|
10 |
6183076 |
0,127 |
C/T |
rs81428333 |
intergenic variant |
|
|
10 |
9242466 |
0,116 |
G/A |
rs81429005 |
intergenic variant |
|
|
11 |
26566153 |
0,112 |
C/A |
rs81430742 |
intergenic variant |
|
|
12 |
47832992 |
0,107 |
C/A |
rs81346419 |
intron variant |
SLC43A2 |
|
13 |
174517355 |
0,153 |
C/T |
rs80967200 |
intergenic variant |
|
|
13 |
175065276 |
0,125 |
T/C |
rs81441358 |
intergenic variant |
|
|
13 |
175654090 |
0,115 |
G/T |
rs340374353 |
intron variant |
ROBO1 |
|
14 |
12995347 |
0,116 |
A/C |
rs80883141 |
intron variant |
KIF13B |
|
14 |
15118851 |
0,130 |
C/T |
rs81230240 |
intergenic variant |
|
|
14 |
15675985 |
0,116 |
C/T |
rs81450331 |
intergenic variant |
|
|
14 |
16719849 |
0,161 |
C/T |
rs80836401 |
intron variant |
GALNT7 |
|
17 |
910440 |
0,112 |
C/T |
rs345772807 |
intron variant |
LONRF1 |
|
17 |
27971107 |
0,105 |
A/G |
rs81000170 |
intron variant |
ENSSSCG00000028004 |
|
17 |
40688317 |
0,113 |
C/T |
rs318433711 |
intergenic variant |
|
|
18 |
5518202 |
0,130 |
T/C |
rs81321404 |
intron variant |
PRKAG2 |
|
18 |
11653824 |
0,131 |
A/C |
rs325655021 |
intron variant |
CREB3L2 |
Молекулярно-генетические исследования основаны на современных знаниях об особенностях молекулы ДНК, а также биохимических процессов транскрипции и трансляции. Главная их цель заключается в выявлении генных мутаций, приводящих к характерным фенотипическим проявлениям. Генные мутации представляют собой изменение расположения, потерю и приобретение участков ДНК относительно ее линейной последовательности, обнаруживаемой в норме.
В нашем исследовании было установлено четыре варианта нуклеотидных замен: синонимичная замена (synonymoys variant); вариант последовательности, расположенный на 3′-конце гена (downstream gene variant); межгенный вариант (intergenic variant); вариант транскрипта, встречающийся внутри интрона (intron variant).
Изначально считалось, что вариации интронов не влияют на фенотип, но недавние исследования подтвердили, что эффекты этих вариаций интронов могут быть причиной множества генетических заболеваний, которые когда-то считались неуловимыми. Интроны могут давать некоторые преимущества в качестве мутационного буфера в эукариотических геномах, защищая кодирующие последовательности от воздействия случайно возникающих вредных мутаций. Интроны занимают в среднем около 40% от общей длины генов, а это означает, что большинство случайно возникающих мутаций приходится на интронные области и не влияет на последовательности и функции белков [5]. Однако неясно, насколько широко и сильно этот буферный эффект интронных областей может воздействовать на эволюционные преимущества.
Генетические варианты возникают не только в белок-кодирующих последовательностях, многие из них попадают в некодирующие регионы, или межгенные области между двумя генами. Например, было подтверждено, что значительный генетический компонент вовлечен в предрасположенность к различным видам рака. Также установлено, что любой продукт гена реализует свой эффект через множество межгенных взаимодействий (отражающих взаимодействие между белками) [11].
Трансформации, которые не меняют конечную последовательность белка, ранее считались молчаливыми мутациями без каких-либо функциональных последствий. Свежие данные показывают, что синонимичные мутации формируются эволюционным отбором и влияют на другие аспекты биогенеза белка. Достижения в области синтетической биологии предоставили исследователям новые методы для понимания разнообразных ролей синонимичных вариаций. Использование синонимичных кодонов воздействует на множество этапов, процессов транскрипции и трансляции, включая регуляцию скорости и точности трансляции, ко-трансляционное сворачивание, посттрансляционные модификации белка, секрецию и уровень экспрессии. Поэтому изучение функций синонимичных мутаций может стать ключом к раскрытию механизма влияния корреляции между полиморфизмами генов и фенотипами [9].
Процессинг 3'-конца матричной РНК представляет собой хорошо организованный процесс, который включает компоненты транскрипции, сплайсинга и механизма трансляции. Пагубное воздействие агрегатов ядерного поли (А)-связывающего белка в скелетных мышцах животных можно уменьшить с помощью шаперон-направляющих подходов или лечения доксициклином. Также доказано, что USEs, стимулирующие формирование 3′-конца мРНК, могут иметь полезные последствия для разработки безопасных стратегий ретровирусной генной терапии. USE повышают эффективность сигнала poly(A), увеличивают эффективность экспрессии трансгенов и сокращают риск активации потенциально опасных клеточных генов путем снижения транскрипции [11].
Выводы
Вариации генома многогранны и служат объяснением межиндивидуального разнообразия, напрямую или косвенно связанного с наследственными заболеваниями и сложными фенотипами. Использование метода FST для поиска подписей селекции позволило выявить 26 SNP, имеющих ассоциации с образованием шишек на конечностях свиней, 15 из которых были локализованы в генах, в том числе регулирующих воспалительные процессы и различного рода новообразования. Установленные SNP в основном представлены нуклеотидными заменами в интронах и межгенными вариантами, что указывает на влияние данных областей на фенотип свиней.
Работа выполнена при поддержке Российского научного фонда, проект №22-76-10015
Литература
- Романец Т.С. Полиморфизм гена ROBO2 и его связь с состоянием конечностей и откормочными качествами свиней породы дюрок/Т.С. Романец, Ф.С. Бакоев, Н.Г. Дуплий. Journal of Agriculture and Environment, 2022. №8(28):13. DOI: 10.23649/jae.2022.28.8.013. ВАК, Agris.
- Bakoev S., Getmantseva L., Kolosova M., Kostyunina O., Chartier D.R., Tatarinova T.V. PigLeg: Prediction of swine phenotype using machine learning. PeerJ, 2020. 8:e8764. https://doi.org/10.7717/peerj.8764.
- Getmantseva L., Kolosova M., Bakoev F., Zimina A., Bakoev S. Genomic regions and candidate genes linked to capped hock in pig. Life (Basel), 2021. 11(6):510. https://doi.org/10.3390/life11060510.
- Hannula-Jouppi K., Kaminen-Ahola N., Taipale M., Eklund R., Nopola-Hemmi J., Kääriäinen H., Kere J. The axon guidance receptor gene ROBO1 is a candidate gene for developmental dyslexia. PLoS Genet., 2005. 1(4):e50. https://doi.org/10.1371/journal.pgen.0010050.
- Jo B.S., Choi S.S. Introns: The functional benefits of introns in genomes. Genomics Inform., 2015. 13(4):112‒8. https://doi.org/10.5808/GI.2015.13.4.112.
- Kolosov A., Getmantseva L., Kolosova M., Romanets T., Bakoev N., Romanets E., Bakoeva I., Kostyunina O., Prytkov Y., Tretiakova O., Bakoev S. Investigation of the genetic architecture of pigs subjected to breeding intensification. Genes (Basel), 2022. 13(2):197. https://doi.org/10.3390/genes13020197.
- Li Y., Zeng C., Hu J., Pan Y., Shan Y., Liu B., Jia L. Long non-coding RNA SNHG7 acts as a target of MIR34A to increase GALNT7 level and regulate PI3K/AKT/mTOR pathway in colorectal cancer progression. J. Hematol. Oncol., 2018. 11(1):89. https://doi.org/10.1186/s13045-018-0632-2.
- Lorenzo-Martín L.F., Menacho-Márquez M., Fernández-Parejo N., Rodríguez-Fdez S., Pascual G., Abad A., Crespo P., Dosil M., Benitah S.A., Bustelo X.R. The Rho guanosine nucleotide exchange factors VAV2 and VAV3 modulate epidermal stem cell function. Oncogene, 2022. 41:3341–3354. https://doi.org/10.1038/s41388-022-02341-7.
- Wang C., Wang S., Liu S., Cheng Y., Geng H., Yang R., Feng T., Lu G., Sun X., Song J. Synonymous mutations of porcine IGF1R extracellular domain affect differentiation and mineralization in MC3T3-E1 cells. Frontiers in Cell and Developmental Biology, 2020. Vol. 8. P. 623. https://doi.org/10.3389/fcell.2020.00623.
- Wang X., Ran X., Niu X., Huang S., Li S., Wang J. Whole genome sequence analysis reveals selection signatures for important economic traits in Xiang pigs. Sci. Rep., 2022. 12:11823. https://doi.org/10.1038/s41598-022-14686-w.
- Zou H., Wu L.X., Tan L., Shang F.F., Zhou H.H. Significance of single-nucleotide variants in long intergenic non-protein coding RNAs. Front Cell Dev. Biol., 2020. 8:347. https://doi.org/10.3389/fcell.2020.00347.