В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2023-5-22-26
УДК 636.02/ 636.03/636.4.033
ВАЛИДАЦИЯ ОЦЕНОК ПЛЕМЕННОЙ ЦЕННОСТИ СВИНЕЙ ПОРОДЫ ДЮРОК ПО ПРИЗНАКАМ КОРМОВОГО ПОВЕДЕНИЯ
П.И. ОТРАДНОВ, младший научный сотрудник, e-mail: deriteronard@gmail.com, Д.М. РУДИЯНОВ, ведущий технолог, e-mail: midiru2393@gmail.com, А.А. БЕЛОУС, кандидат биолог. наук, старший научный сотрудник, e-mail: belousa663@gmail.com, ФГБНУ ФИЦ ВИЖ имени Л.К. Эрнста
Точность оценки племенной ценности животных важна для достижения наибольших темпов генетического прогресса для различных хозяйственно полезных признаков в породных популяциях. В исследовании была проведена сравнительная характеристика повторяемости и взаимосвязанности оценок, полученных в рамках дифференциальных подходов к учету геномной информации.
Ключевые слова: оценки племенной ценности, геномная оценка, автоматические кормовые станции, кормовое поведение, свиньи породы дюрок.
Validation of breeding value estimates for Duroc pigs by feeding behavior traits
P.I. OTRADNOV, junior researcher, e-mail: deriteronard@gmail.com, D.M. RUDIYANOV, leading technologist, e-mail: midiru2393@gmail.com, A.A. BELOUS, candidate of biological sciences, senior researcher, e-mail: belousa663@gmail.com, Federal Research Center for Animal Husbandry named after academy member L.K. Ernst
The accuracy of animal breeding value estimation is important for achieving the maximum genetic progress for various economically important traits in breeding populations. The study conducted a comparative characterization of the repeatability and intercorrelation across estimates obtained using different approaches to incorporating genomic information.
Key words: breeding value, genomic evaluation, automated feeding stations, feeding behavior, Duroc pigs.
Введение
В отрасли свиноводства генетика играет ключевую роль в улучшении экономически важных признаков, таких как качество мяса, рост и репродуктивная способность [1]. Генетическое совершенствование было успешным с использованием традиционных инструментов селекции, таких как метод наилучшего линейного несмещенного прогноза (BLUP) [2]. Тем не менее достигнутый генетический эффект является относительно медленным для признаков, которые считаются дорогостоящими и трудно поддающимися селекции, а именно ‒ измеряемые после выбытия [3‒5].
С другой стороны, в современном свиноводстве все чаще используются методы оценки, основанные на информации о генотипе, ввиду повышения темпов генетического прогресса в популяции, в частности из-за возможности расчёта прогнозных значений племенной ценности до получения сведений о продуктивности животного [1].
Геномная селекция получила широкое признание и успешно применяется в программах селекции животных и растений [6‒8]. Сообщалось, что затраты на разведение молочного скота с использованием геномных методов были на 92% ниже, чем затраты на традиционное тестирование потомства [9].
В настоящее время коэффициент генетического прироста годовой продуктивности американского голштинского молочного скота увеличился примерно с 50% до 100% [10]. На точность и результативность геномной селекции влияет ряд факторов, таких как применяемый аналитический подход, размер референсной популяции, плотность генотипирования и значения наследуемости.
Сегодня для геномной селекции животных чаще всего используются параметрические методы, в основном включающие геномный BLUP (GBLUP) [11], одношаговый GBLUP (ssGBLUP) [12, 13], гребенчатую регрессию (RR) [14], оператора наименьшего абсолютного сокращения и выбора (LASSO) [15] и модели байесовской регрессии с разницей от приоритетного распределения маркерных эффектов [16, 17].
Тем не менее данные линейные модели обычно принимают во внимание только аддитивное (агрегатное) наследование и игнорируют сложные нелинейные отношения, которые могут существовать между SNP-маркерами и фенотипическими значениями (например, эпистаз, доминирование или взаимодействие генотипа с окружающей средой). Кроме того, параметрические методы, как правило, обеспечивают ограниченную гибкость для обработки нелинейных эффектов в многомерных геномных данных, что приводит к большим вычислительным требованиям [18]. Однако исследования показали, что учет нелинейности может повысить способность геномного прогнозирования сложных признаков [19]. Следовательно, необходимо изучить новые подходы для более точной оценки геномной племенной ценности.
Цель исследований – изучить, сравнить и провести валидацию оценок племенной ценности, полученных разными методами.
Материалы и методы исследований
Материалом для исследования были данные первичного учета признаков кормового поведения 1417 хряков породы дюрок 2017‒2020 годов рождения, являвшихся потомками 120 хряков и 499 свиноматок и проходивших откорм на автоматизированных кормовых станциях. 783 особи были генотипированы на ДНК-чипе Porcine GGP HD (платформа GeneSeek Genomic Profiler, Neogene, США), содержащим приблизительно 70 тыс. SNP. Анализировались следующие признаки: среднесуточное потребление корма (ADFI, г), число посещений кормовой станции в сутки (NVD, шт.), среднее время посещения кормовой станции (TPV, мин.), среднее время, проведенное на кормовой станции в сутки (TPD, мин.), скорость потребления корма (FR, г/мин.), среднее количество потребленного корма за посещение (FPV, г).
Характеристики выборки представлены в таблице 1.
Таблица 1. Описательная характеристика исследованных признаков в выборке
|
Показатель/Признак |
ADFI, г |
FPV, г |
NVD, шт. |
TPD, мин. |
TPV, мин. |
FR, г/мин. |
|
Среднее |
2408,36 |
310,92 |
9,10 |
71,07 |
9,34 |
35,58 |
|
Стандартное отклонение |
347,52 |
134,63 |
2,84 |
12,86 |
4,14 |
7,63 |
|
Минимум |
1140,92 |
82,28 |
3,50 |
39,81 |
2,25 |
16,56 |
|
Максимум |
3951,19 |
798,94 |
27,35 |
139,91 |
25,98 |
74,49 |
|
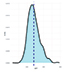
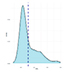
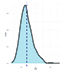
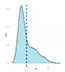
Распределение |
|
|
|
|
|
|
Так, большинство исследованных признаков (ADFI, NVD, TPD, FR) были близки к нормальному распределению. Признаки TPV и FPV были распределены логнормально. Как правило, для подобных случаев дополнительные преобразования не требуются.
Оценка племенной ценности свиней проводилась с применением методологий BLUP Animal Model (AM), GBLUP, ssGBLUP AM и wssGBLUP AM. Для оценки влияния случайных средовых факторов на исследуемые признаки был сформирован рандомизированный эффект DFSM («дата постановки на откорм ‒ номер кормовой станции ‒ возраст в неделях»). Подобная классификация в рамках используемой методологии применяется для определения изменчивости, вызванной случайными эффектами среды и исключения ее воздействия на итоговую оценку племенной ценности животного. В ходе исследования было создано 277 групп DFSM.
Оценка влияния факторов в линейной модели производилась с помощью библиотеки lme4 версии 1.1-32 в программной среде R версии 4.2.3. Модель формировалась по двум критериям – объяснимость воздействия факторов на признак и достоверность влияния факторов α≥0,95.
Таким образом, смешанные математические модели оценки признаков имели вид:

где μ – популяционное среднее, DFSM – рандомизированный эффект среды, BW0 – фиксированный эффект живой массы при постановке на откорм, a – рандомизированный генетический эффект животного (собственно, оценка племенной ценности), e – эффект неучтенных в модели факторов, b1-b3 – фиксированные коэффициенты регрессии оцениваемого признака на соответствующие эффекты.
В общем матричном виде уравнение смешанной модели BLUP AM решается, исходя из уравнения:
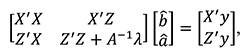
где X, Z – матрицы распределения наблюдений (записей о фенотипах в группах эффектов), X’, Z’ – транспонированные матрицы X и Z, A-1 – обратная матрица родства, 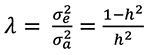 – степень влияния на признак негенетических эффектов,
– степень влияния на признак негенетических эффектов, 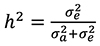 – коэффициент наследуемости.
– коэффициент наследуемости.
В рамках методологии GBLUP обратная матрица родства A-1 заменяется обратной матрицей геномного сходства G-1:
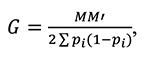
где M – матрица содержания SNP-файла (закодированная зиготность SNP, где 0 – гомозиготность AA, 1 – гетерозиготность AB, 2 – гомозиготность BB, 5 – данные об SNP отсутствуют), M’ – транспонированная матрица M, pi=(1-α), где α – достоверность ассоциации i-го SNP с фенотипическими значениями оцениваемого признака.
Методология ssGBLUP AM предполагает объединение матриц A-1 и G-1 в гибридную матрицу H-1:
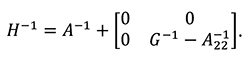
WssGBLUP AM включает дополнительный шаг вычисления индивидуальных весов для каждого SNP, имплементированный в вычисление матрицы геномного родства (VanRaden, 2008):
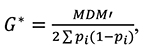
где D – матрица весов SNP ‒ 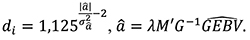
Подытоживая вышесказанное, полноту источников информации о животных в разных подходах к линейной оценке в рамках методологии BLUP можно представить в виде сравнительной таблицы (табл. 2).
Таблица 2. Сравнительная характеристика учитываемых источников информации о животных в рамках методологии BLUP
|
Информация/Метод |
BLUP |
GBLUP |
ssGBLUP |
wssGBLUP |
|
Фенотип |
Наблюдаемые значения признаков |
Наблюдаемые значения признаков |
Наблюдаемые значения признаков |
Наблюдаемые значения признаков |
|
Родственные связи |
Родословная |
Геномное родство |
Родословная, геномное родство |
Родословная, геномное родство |
|
Генотип |
‒ |
Учет степени родства, оценка SNP как агрегатного фактора |
Учет степени родства, оценка SNP как агрегатного фактора |
Учет степени родства, индивидуальная оценка SNP |
Таким образом, наибольшей полнотой информации о животном характеризуется подход wssGBLUP и ssGBLUP, однако GBLUP может использоваться для оценки племенной ценности в отсутствие данных о родословной, а также как индикатор наличия в ней ошибок. Степень повторяемости между оценками BLUP и GBLUP, исходя из методологии, должна быть тем выше, чем больше совпадает степень родства по родословной и генотипу. Кроме того, стоит принимать во внимание, что геномная компонента в GBLUP и ssGBLUP не учитывает различия во влиянии каждого отдельного SNP на фенотип животного, что исправляется в wssGBLUP, но наличие ошибок в родословной все еще может вносить искажения в итоговый результат.
Применение геномных данных к описанным моделям в рамках задействованного методологического аппарата, а также расчет оценок племенной и геномной ценности были реализованы посредством программного обеспечения семейства BLUPF90 с использованием среды разработки RStudio в качестве командной строки.
Валидация полученных оценок в рамках каждого метода осуществлялась в три этапа.
1. Определение достоверности полученных оценок в рамках каждого метода. Расчет показателя достоверности осуществлялся по формуле:
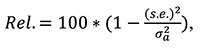
где s.e. – стандартная ошибка оценки племенной ценности, 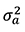 – аддитивная генетическая варианса признака.
– аддитивная генетическая варианса признака.
2. Обособление животных со значением достоверности оценок, превышающим среднее значение достоверности по выборке, в отдельные субвыборки для каждого признака.
3. Переоценка животных в рамках сформированных субвыборок.
4. Определение сходимости полученных в рамках субвыборок оценок путем расчета их коэффициента корреляции с соответствующими им оценками из исходной выборки. Коэффициент корреляции определялся по формуле Пирсона:
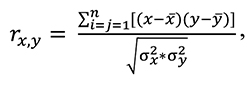
где,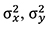 – вариансы x – исходного и y – валидационного массивов оценок.
– вариансы x – исходного и y – валидационного массивов оценок.
Результаты исследований и обсуждение
В ходе исследования были рассчитаны оценки племенной ценности выборки животных на основе четырех подходов в рамках методологии BLUP, определен уровень достоверности результатов для каждого метода, сформирована валидационная субвыборка для каждого признака. Результаты представлены в таблице 3.
Таблица 3. Результаты оценки племенной ценности различными подходами BLUP
|
Признак |
ADFI, г |
FPV, г |
NVD, шт. |
TPD, мин. |
TPV, мин. |
FR, г/мин. |
|
BLUP (n=1417) |
||||||
|
Разброс значений оценки (минимум… максимум) |
-237,24… +257,39 |
-70,38… +67,68 |
-2,82… +4,33 |
-237,24… +257,39 |
-1,49… +1,41 |
-2,62… +2,94 |
|
Средняя достоверность |
27,79 |
33,05 |
37,45 |
27,80 |
26,23 |
25,65 |
|
Объем субвыборки для валидации |
439 |
487 |
456 |
456 |
435 |
434 |
|
Коэффициент наследуемости |
0,309 |
0,395 |
0,471 |
0,309 |
0,284 |
0,275 |
|
GBLUP (n=783) |
||||||
|
Разброс значений оценки (минимум… максимум) |
-123,04… 132,72 |
-85,12… 66,87 |
-0,78… 0,82 |
-7,01… 7,54 |
-1,12… 1,06 |
-1,03… 1,14 |
|
Средняя достоверность |
23,63 |
42,13 |
32,28 |
31,26 |
26,10 |
22,45 |
|
Объем субвыборки для валидации |
439 |
487 |
456 |
456 |
435 |
434 |
|
Коэффициент наследуемости |
0,143 |
0,393 |
0,250 |
0,237 |
0,176 |
0,132 |
|
ssGBLUP (n=1417) |
||||||
|
Разброс значений оценки (минимум… максимум) |
-165,85… 187,74 |
-63,58… 54,78 |
-2,85… 4,39 |
-6,69… 6,81 |
-1,3… 1,23 |
-2,52… 2,77 |
|
Средняя достоверность |
25,65 |
32,82 |
41,79 |
25,48 |
26,02 |
28,17 |
|
Объем субвыборки для валидации |
437 |
463 |
497 |
437 |
438 |
449 |
|
Коэффициент наследуемости |
0,222 |
0,326 |
0,478 |
0,219 |
0,226 |
0,256 |
|
wssGBLUP (n=1417) |
||||||
|
Разброс значений оценки (минимум… максимум) |
-235,59… 223,16 |
-79,95… 67,69 |
-3,23… 5,03 |
-9,59… 8,45 |
-1,67… 1,59 |
-3,1… 3,35 |
|
Средняя достоверность |
31,09 |
37,35 |
45,83 |
31,16 |
30,57 |
32,73 |
|
Объем субвыборки для валидации |
459 |
475 |
520 |
458 |
458 |
464 |
|
Коэффициент наследуемости |
0,300 |
0,400 |
0,551 |
0,301 |
0,291 |
0,325 |
Первое, что обращает на себя внимание, – различающиеся предельные значения полученных массивов оценок для каждого из признаков на основе всех примененных методологий. Возникающие различия в первую очередь обусловлены качеством родословной оцениваемых особей. В гипотетических условиях идеального учета происхождения значения должны претерпевать минимальные изменения, связанные с флуктуациями в матрице родства. Добавление данных о геномном родстве и весов SNP, с учётом методологии формирования матрицы H-1, в описанном контексте не спровоцирует существенных изменений. Однако очевидно, что в исследованной выборке первичный зоотехнический учет не лишен элемента ошибки, чем вызвана как разница в значениях оценок племенной ценности, так и избыточное генетическое разнообразие выборки, выраженное в коэффициентах наследуемости.
Взаимосвязанность оценок каждого признака была определена у генотипированных животных как у имеющих оценки племенной ценности, полученных с применением всех задействованных методов, представлена в таблице 4.
Таблица 4. Взаимосвязь оценок племенной ценности, полученных с применением разных подходов
|
Оценка племенной ценности признака |
BLUP AM | GBLUP |
BLUP AM | wssGBLUP |
ssGBLUP | BLUP AM |
GBLUP | ssGBLUP |
GBLUP | wssGBLUP |
ssGBLUP | wssGBLUP |
|
ADFI |
0,81 |
0,86 |
0,84 |
0,98 |
0,99 |
0,97 |
|
FPV |
0,84 |
0,86 |
0,85 |
0,99 |
0,99 |
0,99 |
|
NVD |
0,66 |
0,86 |
0,85 |
0,78 |
0,99 |
0,76 |
|
TPD |
0,77 |
0,79 |
0,77 |
0,99 |
0,99 |
0,98 |
|
TPV |
0,78 |
0,82 |
0,81 |
0,99 |
0,99 |
0,98 |
|
FR |
0,64 |
0,80 |
0,78 |
0,89 |
0,99 |
0,86 |
|
Минимум… максимум |
0,64…0,84 |
0,79…0,86 |
0,77…0,85 |
0,78…0,99 |
0,99…0,99 |
0,76…0,99 |
Взаимосвязь оценок племенной ценности, выраженная в их корреляциях, позволяет судить о том, насколько ранжирование оцененных животных в рамках каждого массива будет совпадать – иными словами, какова вероятность того, что животное, бывшее лучшим при оценке одним методом, останется лучшим при оценке другим. Уровни взаимосвязей варьировали в пределах 0,64…0,99, что соответствует среднему и высокому уровню коэффициента. Самым низким уровнем корреляций с другими методами характеризовался подход BLUP Animal Model (r=0,64…0,86). Самый низкий уровень был свойственным для пары BLUP AM | GBLUP по признакам NVD (r=0,66) и FR (r=0,64).
Подобные значения свидетельствуют об отсутствии согласованности между методами, вызванным недостатками первичного учета, о которых говорилось ранее. Повышение значений взаимосвязей при добавлении информации о родстве (ssGBLUP и wssGBLUP) увеличивает сходимость оценок на 0,02...0,16 для разных признаков, однако оценки все еще далеки от единообразия.
Взаимосвязи оценок в паре GBLUP | ssGBLUP для ряда признаков характеризовались значениями, близкими к единообразию (признаки ADFI, FPV, TPD, TPV; r=0,98…0,99), однако признаки NVD (r=0,78) и FR (r=0,89) не демонстрировали единообразия. В паре GBLUP | wssGBLUP все признаки были взаимосвязаны на уровне r=0,99, что ввиду учета большего числа источников информации в wssGBLUP дает основание опираться именно на этот метод при оценке племенной ценности. В то же время повторяемость оценок в паре ssGBLUP | wssGBLUP для признаков NVD и FR также была ниже, чем для остальных признаков, что, опять же, объясняется искажениями, вносимыми в оценку ошибками в родословной.
Для выявления повторяемости результатов в рамках применения одного метода была проведена валидация результатов оценки у наиболее достоверно оцененных животных. Результаты представлены в таблице 5.
Таблица 5. Результаты валидационного теста оценок племенной ценности, полученных разными методами
|
Признак/Метод |
BLUP |
GBLUP |
ssGBLUP |
wssGBLUP |
|
ADFI |
0,85 |
0,92 |
0,88 |
0,89 |
|
FPV |
0,94 |
0,91 |
0,87 |
0,88 |
|
NVD |
0,83 |
0,95 |
0,79 |
0,80 |
|
TPD |
0,83 |
0,93 |
0,90 |
0,89 |
|
TPV |
0,89 |
0,91 |
0,87 |
0,90 |
|
FR |
0,70 |
0,91 |
0,75 |
0,79 |
|
Минимум… максимум |
0,70…0,94 |
0,91…0,95 |
0,75…0,90 |
0,79…0,90 |
Результаты валидации оценок племенной ценности демонстрируют наибольшую сходимость значений для метода GBLUP. Это является дополнительным свидетельством в пользу версии о наличии ошибок в первичных данных, в частности в родословной; ssGBLUP и wssGBLUP, в свою очередь, демонстрируют повышение величины наименьшего значения сходимости результатов до 0,75 и 0,79 соответственно. Подобное косвенно подтверждает наличие искажений, вносимых расхождениями между родословной и родством, основанном на данных SNP-генотипирования. Это также соотносится со значениями коэффициентов корреляции между оценками, полученными в рамках различных подходов, представленными в таблице 4.
Заключение
Продемонстрированные различными методами уровни сходимости (повторяемости) результатов свидетельствуют о существенной доле влияния первичных данных на несмещенность оценок племенной ценности, получаемых применением различных подходов. Так, повторяемость оценок племенной ценности с применением классического подхода (BLUP AM), показывает значения на уровне 0,70…0,94, для подхода, учитывающего только геномное родство (GBLUP), – 0,91…0,95, комбинированного родства (ssGBLUP) дает сходимость в пределах 0,75…0,90, учет комбинированного родства и индивидуального веса маркера (wssGBLUP) – 0,79…0,90.
Это свидетельствует о наличии расхождений в степенях геномного родства и классического, основанного на родословной, что подтверждается значениями взаимосвязи оценок, полученных с применением BLUP AM и GBLUP (0,64…0,84). Повышение уровня повторяемости оценок с использованием ssGBLUP и wssGBLUP вызвано тем, что в рамках методологии задействуется массив SNP-данных в качестве дополнительного источника информации о животных. Так, ssGBLUP, учитывая данные SNP-генотипирования, включает в модель маркеры как равновесные компоненты, а в wssGBLUP каждому из маркеров присваивается вес, основанный на его индивидуальных оценках.
Таким образом, использование wssGBLUP видится перспективной методологией оценки племенной ценности в условиях налаженной системы учета происхождения животных. Также видны перспективы применения методического подхода wssGBLUP как ассоциированного с методологией GWAS.
Исследования выполнены в рамках Государственного задания Минобрнауки России №FGGN-2022-0007
Литература
- Badke Y.M., Bates R.O., Ernst C.W., Fix J. and Steibel J.P. Accuracy of estimation of genomic breeding values in pigs using low density genotypes and imputation. G3, 2014. 4:623–631. DOI: 10.1534/g3.114.010504.
- Henderson C.R. Best linear unbiased estimation and prediction under a selection model. Biometrics, 1975. 31:423–447. DOI: 10.2307/2529430.
- Dekkers J.C., Mathur P.K. and Knol E.F. Genetic of the pig improvement//The Genetics of the Pig/eds A. Ruvinski and M.F. Rothschild 390. Wallingford, UK: CAB International, 2010. DOI: 10.1079/9781845937560.0390.
- Wolc A., Arango J., Settar P., Fulton J.E., O’Sullivan N.P., Preisinger R. et al. Persistence of accuracy of genomic estimated breeding values over generations in layer chickens. Genet. Sel. Evol., 2011. 43:23.
- Miar Y., Plastow G. and Wang Z. Genomic selection, a new era for pork quality Improvement. Springer Sci. Rev., 2015. 3:27–37. DOI: 10.1007/s40362-015-0029-3.
- De Roos A.P., Schrooten C., Veerkamp R.F., van Arendonk J.A. Effects of genomic selection on genetic improvement, inbreeding, and merit of young versus proven bulls. J. Dairy Sci., 2011. 94(3):67‒1559.
- Hayes B.J., Bowman P.J., Chamberlain A.J., Goddard M.E. Genomic selection in dairy cattle: Progress and challenges. J. Dairy Sci., 2009. 92(2):43‒433.
- Heffner E.L., Jannink J.L., Sorrells M.E. Genomic selection accuracy using multifamily prediction models in a wheat breeding program. Plant Genome, 2011. 4(1):65–75.
- Schaeffer L.R. Strategy for applying genome-wide selection in dairy cattle. J. Anim. Breed Genet., 2006. 123(4):23‒218.
- García-Ruiz A., Cole J.B., VanRaden P.M., Wiggans G.R., Ruiz-López F.J., Van Tassell C.P. Changes in genetic selection differentials and generation intervals in US Holstein dairy cattle as a result of genomic selection. Proc. Natl. Acad. Sci. USA, 2016. 113(28):E3995–4004.
- Van Raden P.M. Efficient methods to compute genomic predictions. J. Dairy Sci., 2008. 91(11):23‒4414.
- Misztal I., Legarra A., Aguilar I. Computing procedures for genetic evaluation including phenotypic, full pedigree, and genomic information. J. Dairy Sci., 2009. 92(9):55‒4648.
- Christensen O.F., Lund M.S. Genomic prediction when some animals are not genotyped. Genet. Sel. Evol., 2010. 42:2.
- Whittaker J.C., Thompson R., Denham M.C. Marker-assisted selection using ridge regression. Genet. Res., 2000. 75(2):52‒249.
- Tibshirani R. Regression shrinkage and selection via the lasso: A retrospective. J. R. Stat. Soc., 2011. 73(3):82‒273.
- Meuwissen T.H.E., Hayes B.J., Goddard M.E. Prediction of total genetic value using genome-wide dense marker maps. Genetics, 2001. 157(4):29‒1819.
- Habier D., Fernando R.L., Kizilkaya K., Garrick D.J. Extension of the Bayesian alphabet for genomic selection. BMC Bioinform, 2011. 12:186.
- Varona L., Legarra A., Toro M.A., Vitezica Z.G. Non-additive effects in genomic selection. Front. Genet., 2018. 9:78.
- Gianola D., Campos G., González-Recio O., Long N., Wu X.L. Statistical learning methods for genome-based analysis of quantitative traits. In: Proceedings of the 9th World Congress on Genetics Applied to Livestock Production. Leipzig, Germany, 2010. CD-ROM Communication 0014.