В 2025 году журналу
ИСПОЛНИТСЯ
95 ЛЕТ!
ЧИТАЙТЕ
Материалы к юбилею >>>

РУБРИКИ
 Племенное дело |
 Крупный план |
 Актуально |
 Корма |
 Техническая политика |
 Событие |
 Ветеринария |
 Эксклюзивное интервью |
 Статистика |
 Выставки |
Творчество
наших читателей
ЧИТАЙТЕ
Автор С.И. КАЛИНИЧЕНКО
ГИМН СВИНЬЕ >>>
СТАТЬИ ИЗ НОМЕРА
DOI: 10.37925/0039-713X-2024-1-28-30
УДК 636.082.232
ВЛИЯНИЕ ГЕНА TMEM132D НА ВОСПРОИЗВОДИТЕЛЬНЫЕ КАЧЕСТВА СВИНОМАТОК
Е.А. РОМАНЕЦ, аспирант, мл. научный сотрудник, e-mail: lena9258@mail.ru, Т.С. РОМАНЕЦ, кандидат с.-х. наук, ст. преподаватель, e-mail: timofey9258@mail.ru, А.А. АЛЕКСЕЕВ, студент, e-mail: sachaalekseev88@gmail.com, Л.В. ГЕТМАНЦЕВА, доктор биолог. наук, вед. научный сотрудник, e-mail: ilonaluba@mail.ru, ФГБОУ ВО «Донской ГАУ» (п. Персиановский)
Селекция на улучшение воспроизводительных качеств свиноматок позволяет усовершенствовать их репродуктивные характеристики и увеличить объемы продукции. В связи с этим особую актуальность и научную значимость приобретает поиск генетических вариантов, связанных с воспроизводительной продуктивностью свиней.
Ключевые слова: свиньи, TMEM132D, SNP, FST, геномные области, ген.
Effect of TMEM132D gene on reproductive performance of sows
E.A. ROMANETS, postgraduate student, junior researcher, e-mail: lena9258@mail.ru, T.S. ROMANETS, candidate of agricultural sciences, senior lecturer, e-mail: timofey9258@mail.ru, A.A. ALEKSEEV, student, e-mail: sachaalekseev88@gmail.com, L.V. GETMANTSEVA, doctor of biological sciences, leading researcher, e-mail: ilonaluba@mail.ru, Don State Agrarian University (Persianovskiy)
An important task is to increase the efficiency of pig-breeding. Breeding to improve the reproductive qualities of sows will improve their reproductive performance and increase the volume of production. In this regard, the search for genetic variants associated with reproductive performance of pigs is of particular relevance and scientific importance. The aim of the study was to evaluate the effect of TMEM132D gene on reproductive performance of large white pigs and to consider it as a genetic marker for selection and breeding work.
Key words: pigs, TMEM132D, SNPs, FST, genomic regions, gene.
Введение
Улучшение воспроизводительных качеств свиноматок имеет важное значение для развития свиноводства. И генетика играет здесь ключевую роль. В селекционно-племенной работе для отбора свиней с высокой воспроизводительной способностью и передачей этих характеристик будущим поколениям используются гены-маркеры, что сокращает время и ресурсы, затрачиваемые на разведение, и позволяет быстрее достичь желаемых результатов в увеличении плодовитости и массы потомства [1, 2].
Основными показателями воспроизводительной продуктивности свиноматок являются плодовитость (характеризуется такими признаками, как количество поросят при рождении и многоплодие) и масса поросят при рождении (общая масса гнезда и масса одного поросенка).
Хотя эти признаки имеют низкий уровень наследуемости, они связаны с разными генетическими маркерами, которые влияют на процессы воспроизводства [4, 7, 8, 10]. Например, чтобы улучшить репродуктивные качества, исследователи сосредоточились на повышении выживаемости и количества поросят при отъеме путем выявления генетических маркеров, связанных с генами, дифференциально экспрессируемыми на стадии воспроизводства [6].
Проведенные ранее исследования показали перспективность использования гена TMEM132D в качестве гена-маркера воспроизводительных качеств свиней крупной белой породы [3]. На сегодняшний день уже известно, что экспрессия генов семейства TMEM132 имеет широкий спектр влияния при формировании нервной системы различных животных и человека [9].
В случае со свиньями ген TMEM132D может быть связан с адаптацией к стрессу, что является важным при промышленной технологии производства свинины. Исследования также показывают, что у свиней этот ген может быть подвержен искусственному отбору для признаков со сложной архитектурой и иметь связь с репродуктивными признаками [5]. Несмотря на зависимость между стрессом и продуктивными характеристиками у свиней требует дальнейших исследований, ясно, что ген TMEM132D играет важную роль в развитии нервной системы и влияет на продуктивные показатели свиней [11].
Целью исследования является оценка влияния гена TMEM132D на воспроизводительную продуктивность свиней крупной белой породы и рассмотрения его в качестве генетического маркера для селекционно-племенной работы.
Материалы и методы
Исследования проводили на свиньях породы крупная белая (n=232). Для определения генетических вариантов, оказывающих влияние на воспроизводительные качества свиноматок, учитывали показатели по трем опоросам: количество поросят при рождении (TNB_3; гол.), масса одного поросенка при рождении (LWBp_3; кг), многоплодие (NBA_3; гол.) и масса гнезда (LWB_3; кг). Статистическую обработку данных производили в программе R-Studio. Для оценки значимости эффектов генетических вариантов на воспроизводительные качества свиноматок использовали критерий Стьюдента.
Результаты и обсуждение
Параметры описательной статистики признаков воспроизводительной продуктивности у свиноматок (n=232) в исследуемой выборке представлены в таблице 1. Средние показатели воспроизводительных качеств свиноматок в изучаемой выборке находились в пределах стандарта для крупной белой породы свиней. В среднем по трем опоросам количество поросят при рождении и многоплодие у свиноматок составило 13,4 и 12,5 головы, при максимальных показателях ‒ 18,5 и 17,7 голов соответственно. Средняя масса гнеда и масса одного поросенка при рождении составили 14,8 кг и 1,2 кг, при максимальных показателях ‒ 20,5 кг и 1,6 кг.
Таблица 1. Показатели описательной статистики для признаков продуктивности свиней (n=232)
|
Показатель |
Mean |
Min |
Max |
CV |
Range |
SE |
SD |
|
TNB_3 |
13,37 |
7,33 |
18,50 |
14 |
11,17 |
0,123 |
1,87 |
|
NBA_3 |
12,52 |
7,00 |
17,67 |
15,4 |
10,67 |
0,126 |
1,93 |
|
LWB_3 |
14,76 |
7,50 |
20,53 |
16,6 |
13,03 |
0,161 |
2,45 |
|
LWBp_3 |
1,19 |
0,70 |
1,56 |
14,7 |
0,86 |
0,012 |
0,18 |
Примечание. Здесь и далее: Mean – среднее значение, Min – значение минимум, Max – значение максимум, CV – коэффициент вариации, Range – размах между максимальным и минимальным значениями, SE – стандартная ошибка, SD – стандартное отклонение.
После анализа генетической структуры свиней крупной белой породы по гену TMEM132D было обнаружено, что они имеют три генотипа ‒ АА, АС и СС (табл. 2). Наименьшая частота была у гомозиготного генотипа АА (6,9%), частота генотипа АС имела промежуточное значение (40,5%). В исследуемой популяции аллель С был наиболее распространен (72,85%). Соответственно, наибольшая частота наблюдалась у генотипа СС (52,6%), который был выявлен у 122 свиноматок.
Таблица 2. Частота аллелей и генотипов гена TMEM132D у свиней крупной белой породы
|
Выборка, n |
Частота аллелей |
Частота генотипов |
||||||
|
132 |
А |
С |
АА |
АС |
СС |
|||
|
n |
% |
n |
% |
n |
% |
|||
|
27,15 |
72,85 |
16 |
6,9 |
94 |
40,5 |
122 |
52,6 |
|
Таким образом, анализ показал, что ген TMEM132D имеет полиморфный характер у свиней крупной белой породы. Этот полиморфизм дает основание для дальнейших исследований и оценки его влияния на продуктивные показатели свиней с целью использования в качестве ДНК-маркера.
Из таблицы 2 также видно, что частота генотипа АА самая низкая в изучаемой выборке, а частота аллеля А в 2,6 раза меньше частоты аллеля В, при этом наибольшие показатели продуктивности связаны с генотипом АА (табл. 3). Стоит отметить, что у животных с генотипом АА по всем изучаемым признакам размах уровня продуктивности был наименьшим по сравнению с генотипами АС и СС, что указывает на большую однородность животных данного генотипа.
Таблица 3. Показатели описательной статистики генотипов для признаков продуктивности свиней по гену TMEM132D
|
Показатель |
Генотип |
Mean |
Min |
Max |
CV |
Range |
SE |
SD |
|
TNB_3 |
АА |
13,89 |
11 |
17 |
12,79 |
6 |
0,444 |
1,776 |
|
АС |
13,27 |
7,333 |
18,5 |
15,2 |
11,167 |
0,2073 |
2,01 |
|
|
СС |
13,38 |
8 |
17,7 |
13,2 |
9,7 |
0,1596 |
1,763 |
|
|
NBA_3 |
АА |
13,06 |
10 |
16 |
12,57 |
6 |
0,4105 |
1,642 |
|
АС |
12,39 |
7 |
17,5 |
16,9 |
10,5 |
0,2165 |
2,099 |
|
|
СС |
12,56 |
7 |
17,7 |
14,5 |
10,7 |
0,1645 |
1,817 |
|
|
LWB_3 |
АА |
16,44 |
13,25 |
19 |
9,26 |
5,75 |
0,3805 |
1,522 |
|
АС |
15,1 |
9,3 |
19,53 |
16,1 |
10,23 |
0,2504 |
2,428 |
|
|
СС |
14,28 |
7,5 |
20,5 |
17 |
13 |
0,2203 |
2,433 |
|
|
LWBp_3 |
АА |
1,27 |
1,02 |
1,48 |
12,24 |
0,46 |
0,0389 |
0,156 |
|
АС |
1,23 |
0,769 |
1,56 |
14,4 |
0,791 |
0,0183 |
0,178 |
|
|
СС |
1,14 |
0,704 |
1,5 |
14,4 |
0,796 |
0,0149 |
0,165 |
Визуализация уровня воспроизводительной продуктивности изучаемых животных в зависимости от генотипов гена TMEM132D представлена в виде боксплотов (рис. 1‒4). Многоплодие свиноматок с генотипом АА составило 1,27 головы, с генотипом АС и СС ‒ 1,23 и 1,14 головы соответственно (рис. 1). Однако достоверных различий между генотипами по многоплодию не определено.
Масса гнезда при рождении у свиноматок с генотипом АА была на 0,51 кг больше, чем у свиноматок с генотипом СС, и на 0,62 кг больше, чем по генотипу АС (Р<0,001) (рис. 2). Различия были статистически достоверны.
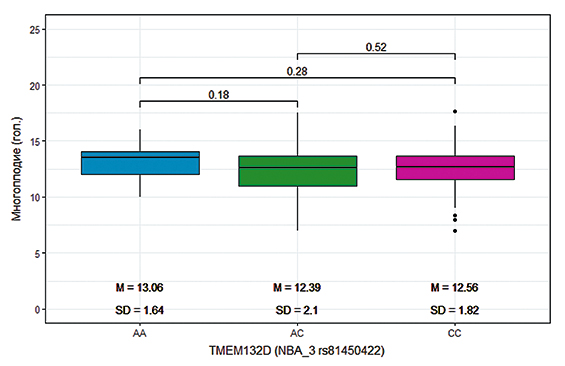
Рис. 1. Многоплодие при различных генотипах
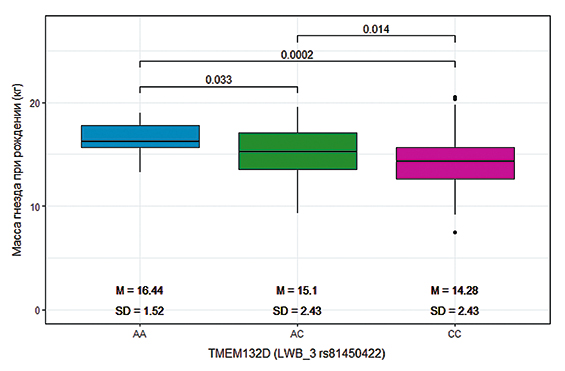
Рис. 2. Масса гнезда при различных генотипах
Свиноматки генотипа АА достоверно превосходили животных с другими генотипами по массе поросят при рождении. У таких свиноматок поросята рождались с более высокой массой. Превосходство по генотипу АА составило 0,13 кг (Р<0,01) в сравнении с генотипом СС (рис. 3).
Количество поросят при рождении у свиноматок генотипа АА составило 13,9 головы, в то время как у свиноматок с генотипом СС – 13,4 головы, однако достоверных различий для количества поросят при рождении, связанных с генотипами, не определено (рис. 4).
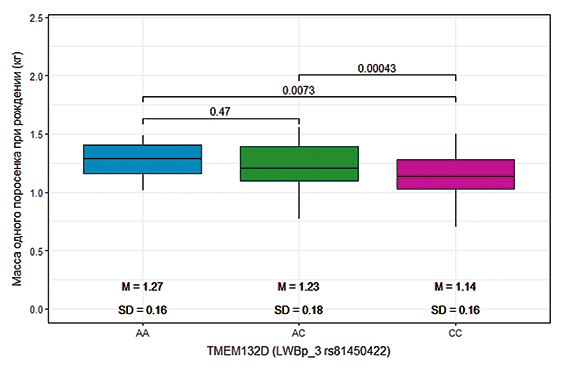
Рис. 3. Масса одного поросенка при рождении при различных генотипах
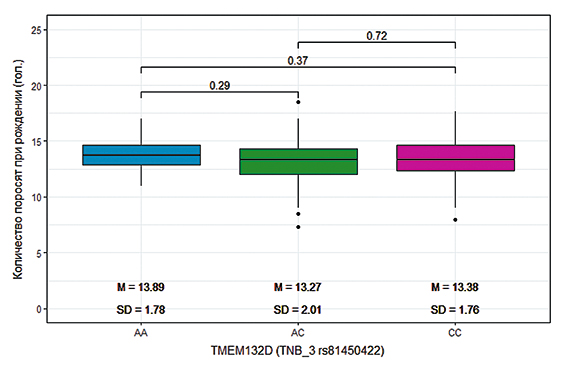
Рис. 4. Количество поросят при рождении при различных генотипах
Заключение
В качестве желательного генотипа по гену TMEM132D в исследуемой выборке установлен генотип AА, наличие которого у свиноматок крупной белой породы относительно других генотипов связано с большим количеством поросят при рождении, многоплодием, массой гнезда, весом одного поросенка при рождении, однако достоверные различия выявлены только по массе поросенка и массе гнезда при рождении.
Ген TMEM132D имеет полиморфный характер у свиней крупной белой породы. Этот полиморфизм дает основание для дальнейших исследований и оценки его влияния на продуктивные показатели свиней с целью использования в качестве ДНК-маркера для селекционно-племенной работы.
Литература
- Колосова М.А. Мировые достижения геномного редактирования в области свиноводства/М.А. Колосова, Е.А. Романец, А.Ю. Колосов, Л.В. Гетманцева. Сибирский вестник сельскохозяйственной науки, 2023. Т. 53. №6. С. 51‒58. DOI: 10.26898/0370-8799-2023-6-6.
- Романец Е.А. Поиск генов-кандидатов селекционно-значимых признаков свиней на основе полногеномного генотипирования/Е.А. Романец, М.А. Колосова, Т.С. Романец, Л.В. Гетманцева. Сборник научных трудов Краснодарского научного центра по зоотехнии и ветеринарии, 2022. Т. 11. №1. С. 321‒323. DOI: 10.48612/sbornik-2022-1-81.
- Романец Е.А. Поиск приоритетных SNP и генов-кандидатов, связанных с массой поросят при рождении свиней крупной белой породы/Е.А. Романец, М.А. Колосова, Т.С. Романец, Л.В. Гетманцева. Сборник научных трудов Краснодарского научного центра по зоотехнии и ветеринарии, 2023. Т. 12. №1. С. 347‒350. DOI: 10.48612/sbornik-2023-1-84.
- 4. Banville M. Genetic parameters for litter size, piglet growth and sow's early growth and body composition in the Chinese-European line Tai-Zumu/M. Banville, J. Riquet, D. Bahon, M. Sourdioux, Canario. Journal of Animal Breeding and Genetics, 2015. Vol. 132. №4. P. 328‒337. https://doi.org/10.1111/jbg.12122.
- Grohmann C.J. Analysis of polygenic selection in purebred and crossbred pig genomes using generation proxy selection mapping/C.J. Grohmann, C.M. Shull, T.E. Crum, C. Schwab, T.J. Safranski, J.E. Decker. Genetics Selection Evolution, 2023. Vol. 55. №1. P. 62. https://doi.org/10.1186/s12711-023-00836-9.
- Lee S.H. Association between polymorphisms of the heart fatty acid binding protein gene and intramuscular fat content, fatty acid composition, and meat quality in Berkshire breed/S.H. Lee, Y.M. Choi, J.H. Choe, J.M. Kim, K.C. Hong, H.C. Park, B.C. Kim. Meat Science, 2010. Vol. 86. №3. P. 794‒800. https://doi.org/10.1016/j.meatsci.2010.06.024.
- Putz A.M. Variance component estimates for alternative litter size traits in swine/A.M. Putz, F. Tiezzi, C. Maltecca, K.A. Gray, M.T. Knauer. Journal of Animal Science, 2015. Vol. 93. №11. P. 5153‒5163. https://doi.org/10.2527/jas.2015-9416.
- Wang X. Genetic determinants of pig birth weight variability/X. Wang, X. Liu, D. Deng, M. Yu, X. Li. BMC Genetics, 2016. Vol. 17. №1. P. 41‒48. https://doi.org/10.1186/s12863-015-0309-6.
- Wang X. The C.elegans homolog of human panic-disorder risk gene TMEM132D orchestrates neuronal morphogenesis through the WAVE regulatory complex/X. Wang, W. Jiang, S. Luo, X. Yang, C. Wang, B. Wang, Dang, Y. Shen, D.K. Ma. Molecular Brain, 2021. Vol. 14. №1. P. 1‒14. https://doi.org/10.1186/s13041-021-00767-w.
- Wang Y. Genome-wide association study of piglet uniformity and farrowing interval/Y. Wang, X. Ding, Z. Tan, C. Ning, K. Xing, T. Yang, Y. Pan, D. Sun, C. Wang. Frontiers in Genetics, 2017. Vol. 8. P. 194. https://doi.org/10.3389/fgene.2017.00194.
- Zhang Z. Genome-wide association study for reproductive traits in a Duroc pig population/Z. Zhang, Z. Chen, S. Ye, Y. He, S. Huang, X. Yuan, Z. Chen, H. Zhang, J. Li. Animals, 2019. Vol. 9. №10. P. 732. https://doi.org/10.3390/ani9100732.








